di Nicolina (Nikka) Mastrangelo
L’avvento di una nuova tecnica, la PCR (Polymerase Chain Reaction), ha rivoluzionato lo studio del DNA sotto il profilo genetico e medico, dando un contributo essenziale alla genetica-forense e rendendo possibile l’analisi degli short tandem repeats o STRs, sequenze ripetute più brevi rispetto ai VNTR.
La PCR fu messa a punto da Kary Mullis tra il 1983 e il 1985 che ebbe il genio di riprodurre in provetta ciò che avviene fisiologicamente nelle nostre cellule: la replicazione del DNA. Questa tecnica, però è più potente poiché consente di amplificare di migliaia di volte il tratto di DNA di nostro interesse a partire da quantità molto esigue, anche da una sola copia del nostro corredo genetico e in poco tempo. Quello che si va ad amplificare sono degli specifici tratti di DNA, nel caso delle applicazioni genetico-forense di tratti polimorfici non codificanti, come i VNTR, ma più piccoli, noti anche come microsatelliti o STRs appunto (Butler 2001).
Gli STRs sono costituiti da frammenti ripetuti la cui unità ripetitiva va da 2 a 6 coppie di basi azotate, e rispetto agli altri polimorfismi di lunghezza, sono maggiormente variabili tra gli individui. La tipizzazione di loci STRs (lucus = posizione di un gene/sequenza nella catena di DNA) nei tests di identificazione umana è stata facilitata dalla possibilità di amplificare più loci simultaneamente in un’unica reazione di PCR, multiplex, utilizzando una coppia di primers specifica per ciascun locus.
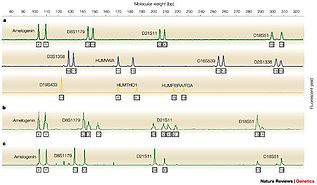
La rilevazione in tempo reale delle varianti di lunghezza dei frammenti amplificati (aventi peso molecolare diverso per il numero variabile di ripetizioni contenute nella regione polimorfa) è consentita grazie alla tecnologia fluorescente accoppiata ad una macchina sequenziatrice in grado di separare e rilevare i diversi frammenti che vengono poi analizzati e tradotti in un grafico, l’elettroferogramma.
La tecnica che oggi viene comunemente utilizzata per la separazione dei frammenti è un’evoluzione dell’elettroforesi ideata da Jeffrey ed è chiamata elettroforesi capillare, distinta proprio perché la separazione avviene in un capillare e non su gel orizzontale. Il metodo è automatizzato: il campione amplificato viene iniettato nel sequenziatore automatico costituito dal capillare contenente un polimero, e viene elettroforeticamente separato (l’elettroforesi in questo caso è più veloce grazie all’alto voltaggio che si può applicare). La fase di separazione viene accoppiata a metodi di rivelazione basati sulla fluorescenza laser indotta che permette di analizzare loci multipli in una singola iniezione capillare, compresi quelli che hanno alleli che si sovrappongono in lunghezza. Alla separazione elettroforetica segue l’analisi e i dati vengono elaborati da un software: il risultato grafico dell’elettroforesi, elettroferogramma, viene restituito come picchi di colore corrispondente a quello del fluorocromo usato per marcare il frammento di interesse.
I microsatelliti, STRs, offrono sostanziali vantaggi:
- sono facilmente amplificabili tramite PCR utilizzando brevi sequenze nucleotidiche (primers) che riconoscono in maniera specifica le regioni fiancheggianti il polimorfismo di interesse;
- possono essere tipizzati con un alto grado di specificità in tempi relativamente brevi;
- la loro moderata variabilità permette una corretta tipizzazione degli alleli mediante confronto con un marcatore di peso molecolare noto (ladder);
- il ristretto range molecolare dei loci STRs, ossia l’intervallo di lunghezza tra l’allele a più basso peso molecolare e quello a più alto peso molecolare, in genere inferiore a 100 paia di basi, consente un’amplificazione omogenea di ciascuno dei due alleli;
- le ridotte dimensioni molecolari delle relative regioni polimorfe permettono l’amplificazione del DNA anche nei casi in cui questo risulti degradato;
- Basso tasso di mutazione;
- Elevata eterozigosità (corrisponde alla quota dei loci di un individuo che sono polimorfi);
- Facilità ad amplificarsi in multiplex.
Da svariati anni sono in commercio kit che permettono l’amplificazione simultanea di numerosi marcatori: oggi esistono kit commerciali, affidabili e validati che consentono di amplificare 24 STRs, comprendenti sia i marker CODIS (Combined DNA Index System) che i marcatori dell’European Standard Set (ESS).
Il CODIS è un programma del FBI di supporto ai database di DNA creati a scopi di giustizia penale, che nel 1997 introdusse 13 loci per l’analisi dei profili (http://www.fbi.gov).
L’ESS è una lista di 7 marcatori genetici proposto dal Consiglio dell’Unione europea da essere utilizzato in Europa da parte degli Stati membri, a fini giudiziari, rispettando i principali obiettivi del Trattato sull’Unione Europea (febbraio 1992) e la risoluzione di questo Consiglio relativa allo scambio di risultati del DNA (giugno 1997) (http://ec.europa.eu).
I polimorfismi STRs sono diffusi anche sui cromosomi sessuali e sul DNA mitocondriale, mtDNA, (ornanelli intracellulari responsabili del metabolismo della cellula e dotati di proprio corredo genetico). Negli ultimi anni si è sviluppato un crescente interesse per questi markers grazie alle loro particolari caratteristiche. Infatti, oltre ovviamente ad indicare il sesso di un individuo, nel caso dei cromosomi X e Y, hanno modalità di trasmissione peculiari che li rendono utili nei casi deficitari di paternità controversa e, più generalmente, nello studio dei rapporti di parentela tra individui, e ancora nelle indagini criminalistiche riguardanti casi di violenza sessuale in cui è fondamentale poter distinguere un profilo maschile in misture di materiale biologico contenente in genere una quantità elevata di DNA della vittima di sesso femminile. Inoltre l’mtDNA diventa essenziale in casi di DNA fortemente degradato poiché presenti nella cellula in un numero piuttosto elevato.
Il cromosoma Y, inoltre, essendo posseduto in singola copia soltanto dagli individui di sesso maschile, ed essendo trasmesso da padre in figlio senza ricombinazione durante la meiosi è utile per gli studi di genetica di popolazione.
In casi più complessi di studio dei rapporti di parentela, ove ovviamente si studiano soggetti di sesso femminile, i markers del cromosoma X si sono rivelati assai utili possedendo un valore più elevato di potere di esclusione rispetto ai markers autosomici. L’utilizzo del cromosoma X in ambito forense è stato fino ad oggi molto limitato per le sue particolari caratteristiche di trasmissione. Infatti, mentre il cromosoma Y in virtù della sua trasmissione in via patrilineare risulta molto vantaggioso poiché permette di studiare in via esclusiva la linea maschile, il cromosoma X viene ereditato in doppia copia dai soggetti di sesso femminile (uno dal padre ed uno dalla madre) ed in singola copia dai soggetti di sesso maschile (solo dalla madre). Questo rende più complessi i calcoli statistici nei casi di paternità e limita inoltre l’indagine ai soli casi in cui il soggetto in studio è di sesso femminile.
L’mtDNA è trasmesso essenzialmente dalla madre ai figli durante la fecondazione e la formazione dello zigote, ha un basso tasso di mutazione e basso potere discriminante. Viene impiegato essenzialmente in studi di identificazione di persone scomparse rispetto ad un parente in linea materna (parenti in linea materna hanno sequenze uguali), di ritrovamenti o disastri di massa.
I marcatori genetici vengono selezionati, approvati e validati per la loro affidabilità in termini di sensibilità dell’analisi e riproducibilità dei risultati, in particolar modo dalla Società Internazionale di Genetica-Forense: ISFG (International Society of Forensic Genetics) nonché da ENFSI, European Network of Forensic Sciences Institutes; EDNAP – European DNA Profiling Group.
I requisiti indispensabili ai quali risponde ciascuno di questi markers comprendono che il polimorfismo sia stabile, cioè che queste forme alternative per lo stesso locus siano mantenuti a un certo livello di equilibrio per periodi di tempo abbastanza lunghi e che essi abbiano frequenze costanti nella popolazione, in accordo con l’equilibrio di Hardy-Weinberg.
Il profiling del DNA trova applicazione in:
- Test di paternità/maternità/parentela
- Casi di violenza sessuale
- Match traccia biologica – individuo sospettato/vittima
- Identificazione di persone scomparse
- Identificazione nei disastri di massa
- Costruzione di banche dati
I campioni da analizzare possono essere di varia natura. A parte il tampone salivare utilizzato per i test in laboratorio, in genere ci si trova a lavorare con campioni particolari, recuperati in scenari legati a eventi criminosi o comunque di interesse forense. Essi comprendono: sangue, liquido seminale, saliva, urina, capelli, denti, ossa, tessuti.
Ai fini investigativi lo scopo è ottenere informazioni identificative da un profilo da associare ad un individuo, per cui l’analisi è volta a stabilire una comparazione.
Bibliografia
Mullis K., Faloona F., Scharf S., Saiki R., Horn G., and Erlich H.: Specific enzymatic amplifcation of DNA in vitro: the polymerase chain reaction, Cold Spring Harbour Symposium on Quantitative Biology 51 1986 263-273;
Butler J.M. Advanced Topics in Forensic DNA Typing: Methodology: Methodology 2011 Academic Press;
Butler JM, Hill CR: Biology and genetics of new autosomal STR loci useful for forensic DNA analysis; Forensic Sci 2012 Rev 24:15;
Butler J.M. The future of forensic DNA analysis. 2015 Phil. Trans. R. Soc;
Tagliabracci 2010. Introduzione alla genetica forense. Indagini di identificazione personale e di paternità, Springer, Italia